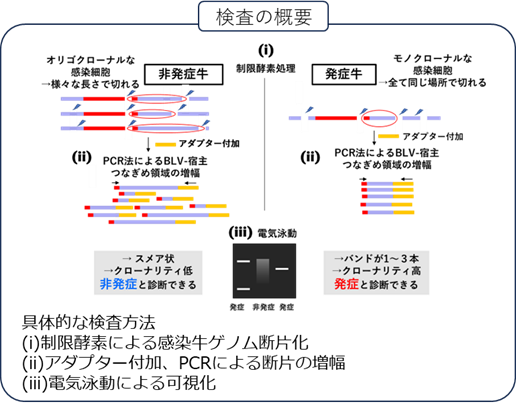
牛伝染性リンパ腫発症を簡易的に検査する、BLV-AFLP法のプロトコールです。
①DNA抽出
感染牛の血液200 ulよりQIAamp DNA Blood Mini Kit (QIAGEN)を用いてDNA抽出
あるいは、
発症牛の組織よりDNeasy Blood and tissue kit (QIAGEN)を用いてDNA抽出
②制限酵素処理
PCRチューブを使用
感染牛血液・腫瘍から抽出したDNA 300ng
Mse I (NEB R0525S 10,000 units/ml) 2 µl
rCuSmartBuffer 2 µl
DEPC water (up to 20 µl)
37℃5分 サーマルサイクラーにて反応させる。
(total volume 20ulを確認)
③エンドプレップ
制限酵素処理後のチューブに以下の試薬を加える。
(ice boxなどで冷やしながら行う、以降の反応も同様)
NEBNext Ultra II DNA Library Prep Kit for Illumina (#E7645)を使用
NEBNext Ultra II End Prep Enzyme Mix (緑の蓋) 1.2 µl
NEBNext Ultra II End Prep Reaction Buffer(緑の蓋) 2.8 µl
制限酵素処理したDNA 20 µl
(全量24 ulとなる)
10回程度ピペッティングする。
20℃30分→65℃30分→4℃∞
④アダプターライゲーション反応
NEBNext® Multiplex Oligos for Illumina® (Index Primers Set 1) (# E7335)を使用
エンドプレップ反応液(上記のもの) 24 µl
NEBNext Adaptore for illumina (赤い蓋) 1 µl
NEBNext Ultra II Ligation Master Mix (赤い蓋) 12 µl
NEBNext Ligation Enhancer (赤い蓋) 0.4 µl
全量 37.4 µl
※NEBNext Ultra II Ligation Master Mix は数回ピペッティングして混合し、反応液に添加する。
※NEBNext Ultra II Ligation Master Mix とLigation Enhancerは事前に混合しておくことが可能。
アダプターの事前混合は避け、必ず最後に添加する。
10回以上ピペッティング
ヒートリッド(上部の鉄板)の温度設定を切る。
サーマルサイクラーにて、20℃15分
反応液に、USER enzyme 1.2ulを加え、よく混ぜる。全量は38.6 ulとなる。
(USER Enzymeはライブラリー調整キットではなく、インデックスプライマーキットに入っている。)
ヒートリッド(上部の鉄板)の温度を47℃以上に設定。
サーマルサイクラーにて、37℃15分
⑤カラム精製 QIAquick PCR Purification Kitを使用
1. PCRサンプルに5倍容量のBuffer PBを入れ混和する。(38.6 ulの5倍量=193 µl)
2. DNAを結合させるために、サンプルをQIAquickカラムにアプライし、30~60秒間遠心操作する。
3. ろ液は棄てる。同じチューブの上にQIAquickカラムを再度のせる。
4. 洗浄のために750 µlのBuffer PEをQIAquickカラムにアプライする。
5. 30~60秒間遠心操作する。
6. ろ液は棄て、QIAquickカラムを同じコレクションチューブに再度のせる。さらに1分間カラムを遠心操作する。
7. 新しい1.5 mlのマイクロ遠心チューブにQIAquickカラムをのせる。
9. DNAの溶出を行なうために、30 µlのDEPC waterをQIAquickメンブレンの中央に添加し、1分間カラムを放置後、1分間遠心する。
重要:結合したDNAを完全に溶出するために、溶出バッファーをQIAquickメ
ンブレンに直接アプライする。
⑥制限酵素処理
溶出したAdaptor ligated DNA 25 µl
Bcl1-HF (NEB R3160S 20,000 units/ml) 3 µl
rCut Smart buffer 5 µl
Water 17 µl (up to 50 µl)
at 37C for 5 min (total volume 50 µlを確認)
1.5 mlチューブで反応させてもOK
⑦1stPCR
東洋紡のKOD Plus Neoを使用。
プライマーは以下
BLV-AFLP_1stF CTT GCA CCC RCG TTY GTT TC
Index1Primer CAA GCA GAA GAC GGC ATA CGA GAT CGT GAT GTG ACT GGA GTT CAG ACG TGT GCT CTT CCG ATC T
10x Buffer for KOD-Plus Neo (TOYOBO) 5 µl
2mM dNTPs(each) 5 µl
25mM MgSO4 3 µl
10uM BLV-AFLP_1stF (10uM) 1.5 µl
10uM Index1 primer (10uM) 1.5 µl
KOD-Plus Neo 1 µl
Adaptor ligated DNA 5 µl
Water 28 µl (up to 50 µl)
Total 50 µl
サーマルサイクラーは、
94 ℃ 2min
98 ℃ 10sec 30cycles
68 ℃ 43sec
4℃
⑧2ndPCR
東洋紡のKOD Plus Neoを使用。
プライマーは以下
BLV-AFLP_2ndF CTT ACT TTC TGT TTC TCG CGG C
P7_Rev CAA GCA GAA GAC GGC ATA CGA GAT
10x Buffer for KOD-Plus Neo(TOYOBO) 5 µl
2mM dNTPs(each) 5 µl
25mM MgSO4 3 µl
10uM BLV-AFLP_2ndF (10uM) 1.5 µl
10uM p7_Rev (10uM) 1.5 µl
KOD-Pl 1 µl
1st PCR products 1 µl
Total 50ul
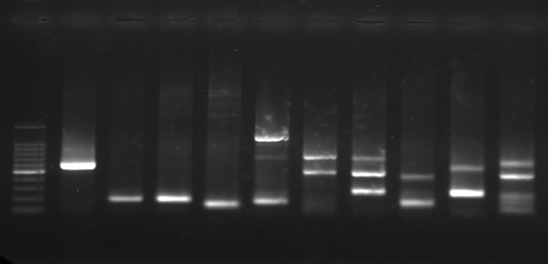
電気泳動
1%ゲル、5000bpラダー(LABTAS LT-10-DM-428S)5 µlを使用。
バンドを切り出し、あるいはクローニングし、シーケンス解析によりインテグレーションサイトを決定することも可能。
スメア状でも発症している場合もある
→制限酵素切断部位がBLV組込み位置に対してPCRで増幅可能な位置にない場合
→腫瘍組織のクローンが血液中に循環しない場合
→散発性のリンパ腫の場合
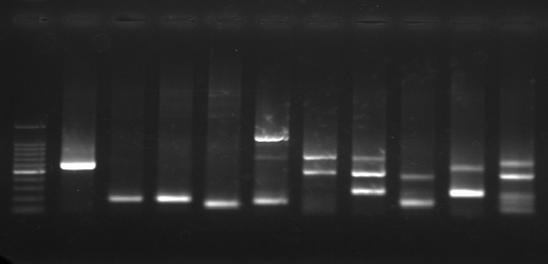
発症すると明瞭な少数のバンド、非発症ではスメア状のバンドが観察されます(写真は全て発症牛)。
詳しくはこちらの論文をご参照ください→10.1128/spectrum.01714-24